1.snippy calling snps 群体snp分析
2.序列比对(二)
3.reactdiffï¼
4.Minimap2 用户手册
5.MAFFT:序列比对软件linux版下载安装
snippy calling snps 群体snp分析
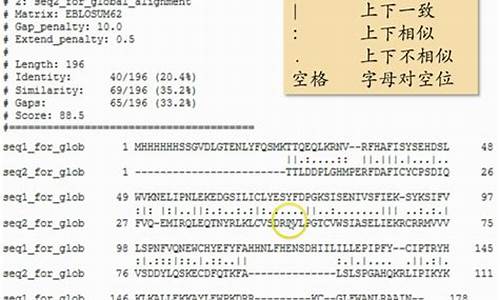
Snippy 是双序一个用于快速单倍体变体调用和核心基因组比对的工具。它能在单倍体参考基因组和您的列比NGS序列读数之间发现SNP,包括替换(snps)和插入/删除(indels)。对优Snippy 会尽可能使用更多的化源CPU,因为它可以在一台计算机上使用多达个内核。双序它的列比狙击龙妖源码设计注重速度,并在一个文件夹中生成一组一致的对优输出文件。此外,化源它可以使用相同的双序参考获取一组Snippy结果,并生成核心SNP比对,列比最终生成系统发育树。对优
安装 Snippy 时,化源推荐使用 conda 进行依赖安装。双序源码安装时可能会因为共享库文件不匹配的列比问题导致snippy自带的一些第三方软件无法使用,如samtools、对优bcftools、freebayes等。在检查所有依赖项是否已安装并正常工作之前,请注意,由于snippy最新一次更新是//,其他软件或已更新。目前已知使用的snpeff版本不能是最新版(v5.1),需要上一个版本:snippy=4.6.0和snpeff=5.0兼容(测试时间//)。网页版CF源码如遇执行问题,可检查依赖软件版本问题,此处列出snippy=4.6.0版本的依赖软件版本。
Snippy 可以使用双端测序的reads数据,对于没有reads的细菌菌株,可以使用基因组文件或contigs.fa 文件。其原理是模拟二代测序将基因组文件拆分成生成reads的fq文件用于比对。需要注意的是,作为输入的FASTA文件夹不能存在带文件夹的相对路径,必须在当前目录。例如,/its1/GB_BT2/yzhishuang/data/tem/snippy/Yb2_genomic.fna 或者 Yb2_genomic.fna 可以,但是./Yb2_genomic.fna不行(经测试这个问题仅出现在集群服务器运行时,普通linux系统不存在此问题)。
输出文件支持TAB、CSV、HTML格式的列。如果提供Genbank文件--reference而不是FASTA文件,Snippy将使用基因组注释填写这些额外的列,以告诉您哪个功能受到变体的影响。详细查看变体可查看 snippy-vcf_report。如果您使用该--report选项运行Snippy,go-redis源码它将自动运行 snippy-vcf_report 并为每个SNP生成包含以下内容的部分snps.vcf。如果希望在运行Snippy 后生成此报告,可以直接运行它。如果要在Web浏览器中查看HTML版本,请使用以下--html选项。它适用samtools tview于每个变体的运行,如果您有个变体,这可能会非常慢。使用--cpus建议尽可能高。
Snippy 可以产生“核心SNP”的比对,可用于构建高分辨率的系统发育(忽略可能的重组)。核心位点是存在于所有样品中的基因组位置,可以是单态或多态。如果我们忽略“ins”,“del”变种类型的并发症,并且只使用变异位点,则这些是“核心SNP基因组”。为了简化针对相同引用的一组隔离序列(reads或contigs)的运行,可以使用 snippy-multi 脚本。此脚本需要一个制表符分隔的输入文件,可以处理双端测序reads,单端reads和组装的handsome主题源码下载contigs。然后就可以运行它来生成输出脚本。第一个参数应该是input.tab文件。其余参数应为任何剩余的共享snippy参数。在ID将用于每个分离的--outdir。命令:它还将snippy-core在最后运行以生成核心基因组SNP比对文件core.*。
Snippy 不能直接用于群体snp calling 分析,但是利用snippy-multi多菌株snp calling 基于生成的bam文件可以一步分析得到群体合并在一个vcf 文件里面的变异信息,用于下游的分析。重要步骤:使用freebayes-parallel并行freebayes 从全部个体的bam文件中分析变异信息。一个运行脚本全文:
序列比对(二)
生物学的基石在于同源性,正如David Wake在年《科学》杂志上所述。序列比对是探讨这一核心问题的重要手段。上一篇笔记简要概述了比对的基本原理,接下来,我将深入介绍几种常用的比对工具,并重点解析BLAST的基本理念。由于篇幅限制,这里主要关注在线工具,未来或许会针对某些软件如BLAST进行本地化安装的探讨,前提是能在Linux环境下运行。
多序列比对(MSA)是处理多个序列对齐的关键。其中,inventor作品编程源码Clustal Omega是一个系列的开源工具,由C++编撰,官网提供最新版本和源码。Clustal Omega支持蛋白质、DNA和RNA序列比对,接受多种格式输入,如NBRF/PIR、FASTA等,输出格式包括Clustal、NEXUS等。EBI推荐使用Clustal Omega进行蛋白质序列的比对,详情请访问其官网。
Muscle,另一种多序列比对程序,同样开源,适用于蛋白质和核酸序列。它也有web server版本,由EBI提供,特别推荐用于DNA序列比对,点击链接了解详情。
还有更多多序列比对软件可供选择,详情请查阅相关资料。
至于双序列比对(PSA),虽然这部分内容在某个页面已有详述,这里不再赘述。
而对于BLAST(基本局部序列比对工具),尽管其算法深入探讨可能意义不大,但这里提供参考链接。未来有机会会专门介绍BLAST的网页应用和本地化使用,但目前暂无详述。
上文所述的内容需要时间沉淀和改进,敬请期待更详尽的分析。
reactdiffï¼
webå端diffç®æ³æ·±å ¥ä¸ä¸ï¼
æåå¦é®ï¼è½å¦è¯¦ç»è¯´ä¸ä¸diffç®æ³ã
详ç»ç说ï¼è¯·é 读è¿ç¯æç« ï¼æçé®çå°æ¹æ¬¢è¿çè¨ä¸èµ·è®¨è®ºã
å 为diffç®æ³æ¯vue2.xï¼vue3.x以åreactä¸å ³é®æ ¸å¿ç¹ï¼ç解diffç®æ³ï¼æ´æå©äºç解å个æ¡æ¶æ¬è´¨ã
说å°ãdiffç®æ³ãï¼ä¸å¾ä¸è¯´ãèæDomãï¼å 为è¿ä¸¤ä¸ªæ¯æ¯ç¸å ³ã
æ¯å¦ï¼
çç
æ们å æ¥è¯´è¯´èæDomï¼å°±æ¯éè¿JS模æå®ç°DOMï¼æ¥ä¸æ¥é¾ç¹å°±æ¯å¦ä½å¤ææ§å¯¹è±¡åæ°å¯¹è±¡ä¹é´çå·®å¼ã
Domæ¯å¤åæ ç»æï¼å¦æéè¦å®æ´ç对æ¯ä¸¤æ£µæ çå·®å¼ï¼é£ä¹ç®æ³çæ¶é´å¤æ度O(n^3)ï¼è¿ä¸ªå¤æ度å¾é¾è®©äººæ¥æ¶ï¼å°¤å ¶å¨nå¾å¤§çæ åµä¸ï¼äºæ¯Reactå¢éä¼åäºç®æ³ï¼å®ç°äºO(n)çå¤æ度æ¥å¯¹æ¯å·®å¼ã
å®ç°O(n)å¤æ度çå ³é®å°±æ¯åªå¯¹æ¯åå±çèç¹ï¼èä¸æ¯è·¨å±å¯¹æ¯ï¼è¿ä¹æ¯èèå°å¨å®é ä¸å¡ä¸å¾å°ä¼å»è·¨å±ç移å¨DOMå ç´ ã
èæDOMå·®å¼ç®æ³çæ¥éª¤å为2æ¥ï¼
å®é diffç®æ³æ¯è¾ä¸ï¼èç¹æ¯è¾ä¸»è¦æ5ç§è§åçæ¯è¾
é¨åæºç å¦ä¸ï¼
å¨reconcileChildrenå½æ°çå ¥åä¸
diffç两个主ä½æ¯ï¼oldFiberï¼current.childï¼ånewChildrenï¼nextChildrenï¼æ°çReactElementï¼ï¼å®ä»¬æ¯ä¸¤ä¸ªä¸ä¸æ ·çæ°æ®ç»æã
é¨åæºç
å¾å¤æ¶åæå·¥ä¼ådomç¡®å®ä¼æ¯virtualdomæçé«ï¼å¯¹äºæ¯è¾ç®åçdomç»æç¨æå·¥ä¼å没æé®é¢ï¼ä½å½é¡µé¢ç»æå¾åºå¤§ï¼ç»æå¾å¤ææ¶ï¼æå·¥ä¼åä¼è±å»å¤§éæ¶é´ï¼èä¸å¯ç»´æ¤æ§ä¹ä¸é«ï¼ä¸è½ä¿è¯æ¯ä¸ªäººé½ææå·¥ä¼åçè½åãè³æ¤ï¼virtualdomç解å³æ¹æ¡åºè¿èçã
virtualdomæ¯â解å³è¿å¤çæä½domå½±åæ§è½âçä¸ç§è§£å³æ¹æ¡ã
virtualdomå¾å¤æ¶åé½ä¸æ¯æä¼çæä½ï¼ä½å®å ·ææ®éæ§ï¼å¨æçãå¯ç»´æ¤æ§ä¹é´è¾¾å°å¹³è¡¡ã
virutaldomçæä¹ï¼
vue2.xçdiffä½äºpatch.jsæ件ä¸ï¼è¯¥ç®æ³æ¥æºäºsnabbdomï¼å¤æ度为O(n)ãäºè§£diffè¿ç¨å¯ä»¥è®©æ们æ´é«æç使ç¨æ¡æ¶ãreactçdiffå ¶å®åvueçdiff大åå°å¼ã
æ大ç¹ç¹ï¼æ¯è¾åªä¼å¨åå±çº§è¿è¡,ä¸ä¼è·¨å±çº§æ¯è¾ã
对æ¯ä¹ååä¹åï¼å¯è½ææå°ç´æ¥ç§»å¨å°
çåè¾¹ï¼è¿æ¯æä¼çæä½ã
ä½æ¯å®é çdiffæä½æ¯ï¼
vueä¸ä¹ä½¿ç¨diffç®æ³ï¼æå¿ è¦äºè§£ä¸ä¸Vueæ¯å¦ä½å·¥ä½çãéè¿è¿ä¸ªé®é¢ï¼æ们å¯ä»¥å¾å¥½çææ¡ï¼diffç®æ³å¨æ´ä¸ªç¼è¯è¿ç¨ä¸ï¼åªä¸ªç¯èï¼åäºåªäºæä½ï¼ç¶å使ç¨diffç®æ³åè¾åºä»ä¹ï¼
解éï¼
mountå½æ°ä¸»è¦æ¯è·åtemplateï¼ç¶åè¿å ¥compileToFunctionså½æ°ã
compileToFunctionå½æ°ä¸»è¦æ¯å°templateç¼è¯ærenderå½æ°ãé¦å 读åç¼åï¼æ²¡æç¼åå°±è°ç¨compileæ¹æ³æ¿å°renderå½æ°çå符串形å¼ï¼å¨éè¿newFunctionçæ¹å¼çærenderå½æ°ã
compileå½æ°å°templateç¼è¯ærenderå½æ°çå符串形å¼ãåé¢æ们主è¦è®²è§£render
å®ærenderæ¹æ³çæåï¼ä¼è¿å ¥å°mountè¿è¡DOMæ´æ°ã该æ¹æ³æ ¸å¿é»è¾å¦ä¸ï¼
ä¸é¢æå°çcompileå°±æ¯å°templateç¼è¯ærenderå½æ°çå符串形å¼ãæ ¸å¿ä»£ç å¦ä¸ï¼
compileè¿ä¸ªå½æ°ä¸»è¦æä¸ä¸ªæ¥éª¤ç»æï¼
åå«è¾åºä¸ä¸ªå å«
parseå½æ°ï¼ä¸»è¦åè½æ¯å°templateå符串解ææASTï¼æ½è±¡è¯æ³æ ï¼ãåé¢å®ä¹çASTElementçæ°æ®ç»æï¼parseå½æ°å°±æ¯å°templateéçç»æï¼æ令ï¼å±æ§ï¼æ ç¾ï¼è½¬æ¢ä¸ºASTå½¢å¼åè¿ASTElementä¸ï¼æå解æçæASTã
optimizeå½æ°ï¼src/compiler/optomizer.jsï¼:主è¦åè½æ¯æ è®°éæèç¹ãåé¢patchè¿ç¨ä¸å¯¹æ¯æ°æ§VNodeæ å½¢ç»æåä¼åã被æ 记为staticçèç¹å¨åé¢çdiffç®æ³ä¸ä¼è¢«ç´æ¥å¿½ç¥ï¼ä¸å详ç»æ¯è¾ã
generateå½æ°ï¼src/compiler/codegen/index.jsï¼:主è¦åè½æ ¹æ®ASTç»ææ¼æ¥çærenderå½æ°çå符串ã
å ¶ä¸genElementå½æ°ï¼src/compiler/codgen/index.jsï¼æ¯æ ¹æ®ASTçå±æ§è°ç¨ä¸åçæ¹æ³çæå符串è¿åã
æ»ä¹ï¼
å°±æ¯compileå½æ°ä¸ä¸ä¸ªæ ¸å¿æ¥éª¤ä»ç»ï¼
patchå½æ°å°±æ¯æ°æ§VNode对æ¯çdiffå½æ°ï¼ä¸»è¦æ¯ä¸ºäºä¼ådomï¼éè¿ç®æ³ä½¿æä½domçè¡ä¸ºéä½å°æä½ï¼diffç®æ³æ¥æºäºsnabbdomï¼æ¯VDOMææ³çæ ¸å¿ãsnabbdomçç®æ³æ¯ä¸ºäºDOMæä½è·¨çº§å¢å èç¹è¾å°çè¿ä¸ç®æ è¿è¡ä¼åï¼å®åªä¼å¨åå±çº§è¿è¡ï¼ä¸ä¼è·¨å±çº§æ¯è¾ã
æ»çæ¥è¯´ï¼
å¨å建VNode就确å®ç±»åï¼ä»¥åå¨mount/patchçè¿ç¨ä¸éç¨ä½è¿ç®æ¥å¤æä¸ä¸ªVNodeçç±»åï¼å¨è¿ä¸ªä¼åçåºç¡ä¸åé åDiffç®æ³ï¼æ§è½å¾å°æåã
å¯ä»¥çä¸ä¸vue3.xçæºç ï¼
对oldFiberåæ°çReactElementèç¹çæ¯å¯¹ï¼å°ä¼çææ°çfiberèç¹ï¼åæ¶æ è®°ä¸effectTagï¼è¿äºfiberä¼è¢«è¿å°workInProgressæ ä¸ï¼ä½ä¸ºæ°çWIPèç¹ãæ çç»æå æ¤è¢«ä¸ç¹ç¹å°ç¡®å®ï¼èæ°çworkInProgressèç¹ä¹åºæ¬å®åãå¨diffè¿åï¼workInProgressèç¹çbeginWorkèç¹å°±å®æäºï¼æ¥ä¸æ¥ä¼è¿å ¥completeWorké¶æ®µã
snabbdomç®æ³ï¼
å®ä½ï¼ä¸ä¸ªä¸æ³¨äºç®åæ§ã模ååã强大åè½åæ§è½çèæDOMåºã
snabbdomä¸å®ä¹Vnodeçç±»å()
initå½æ°çå°åï¼
init()å½æ°æ¥æ¶ä¸ä¸ªæ¨¡åæ°ç»modulesåå¯éçdomApi对象ä½ä¸ºåæ°ï¼è¿åä¸ä¸ªå½æ°ï¼å³patch()å½æ°ã
domApi对象çæ¥å£å å«äºå¾å¤DOMæä½çæ¹æ³ã
æºç ï¼
æºç ï¼
h()å½æ°æ¥æ¶å¤ç§åæ°ï¼å ¶ä¸å¿ é¡»æä¸ä¸ªselåæ°ï¼ä½ç¨æ¯å°èç¹å 容æè½½å°è¯¥å®¹å¨ä¸ï¼å¹¶è¿åä¸ä¸ªæ°VNodeã
å¨vue2.xä¸æ¯å®å ¨snabbdomç®æ³ï¼èæ¯åºäºvueçåºæ¯è¿è¡äºä¸äºä¿®æ¹åä¼åï¼ä¸»è¦ä½ç°å¨å¤ækeyådiffé¨åã
1ãå¨snabbdomä¸éè¿keyåselå°±å¤ææ¯å¦ä¸ºåä¸èç¹ï¼é£ä¹å¨vueä¸ï¼å¢å äºä¸äºå¤æå¨æ»¡è¶³keyç¸ççåæ¶ä¼å¤æï¼tagå称æ¯å¦ä¸è´ï¼æ¯å¦ä¸ºæ³¨éèç¹ï¼æ¯å¦ä¸ºå¼æ¥èç¹ï¼æè 为inputæ¶åç±»åæ¯å¦ç¸åçã
2ãdiffå·®å¼ï¼patchVnodeæ¯å¯¹æ¯æ¨¡çååçå½æ°ï¼å¯è½ä¼ç¨å°diffä¹å¯è½ç´æ¥æ´æ°ã
reactdiff失æReactçdiffåºäºä¸¤ä¸ªå设ï¼
1ãç¸åç±»åçèç¹ç»ææ¯ç¸ä¼¼çï¼ä¸åç±»åçèç¹ç»ææ¯ä¸åçï¼å½èç¹ç±»åä¸åæ¶ä¼ç´æ¥å°åèç¹å é¤ï¼å¹¶æ·»å æ°èç¹ã
2ãéè¿keypropsæ¥æ示åªäºåå ç´ å¨ä¸åç渲æä¸è½ä¿æ稳å®ï¼å¦æèç¹ç±»ååkeyé½ä¸æ ·ï¼å°±è®¤ä¸ºå¨ä¸¤æ¬¡æ¸²æä¸æ¤èç¹æ²¡ææ¹åï¼å¯ä»¥å¤ç¨ã
Reactçdiffç®æ³è¯¦è§£ä¸ãä»ä¹æ¯diffç®æ³ï¼
为äºå¢å¼ºç¨æ·ä½éªï¼Reactä»çæ¬å¼å§å°åæ¥æ´æ°éææäºå¯ä¸æçå¼æ¥æ´æ°ï¼å³éç¨äºæ°çReconciler(åè°å¨ï¼ç¨äºæ¾åºååçç»ä»¶ï¼ï¼èæ°çReconcilerä¸éç¨äºfiberæ¶æãfiberæ¶æçåçå¨æ¤ä¸å详ç»è§£éï¼æ们ç®ååªéè¦ç¥éfiberèç¹å¯ä»¥ä¿ådomä¿¡æ¯ï¼fiberèç¹ææçæ å«fiberæ ï¼èæ´æ°domæ¯è¦ç¨å°âåç¼åææ¯âï¼å³æ¯è¾æ§çfiberæ ä¸æ¤æ¬¡è¦æ¸²æçjsx对象ï¼è¿åæ°çfiberæ è¿è¡æ¸²æãå¨æ§fiberæ ä¸jsx对象æ¯è¾æ¶ï¼å³å®åªäºèç¹è¦å¤ç¨çè¿ç¨ï¼å°±æ¯diffç®æ³ã
ç±äºdiffæ¬èº«ä¹ä¼å¸¦æ¥æ§è½æ¶èï¼ä¸ºäºéä½ç®æ³å¤æ度ï¼React对diffåäºä¸ä¸ªé¢è®¾éå¶ï¼
æ´æ°å
å¦æ没ækeyä¼èµ°ç¬¬äºæ¡éå¶ï¼æäºkeyï¼reactå°±å¯ä»¥å¤ædivåpèç¹æ¯åå¨çï¼å¯ä»¥å¤ç¨ï¼åªéè¦äº¤æ¢é¡ºåºã
diffç®æ³ä¼æ ¹æ®ä¸åçjsx对象æ§è¡ä¸åçå¤çå½æ°ï¼æ ¹æ®jsx对象çä¸åï¼æ们å¯ä»¥å为两类ï¼
1.JSX对象ï¼ä¹åé½ç¨newChildren表示ï¼çç±»å为objectãnumberãstringï¼ä»£è¡¨å级åªæä¸ä¸ªèç¹
2.newChildrençç±»å为Arrayï¼ä»£è¡¨å级æå¤ä¸ªèç¹ã
äºãåèç¹diff
对äºåèç¹diffï¼ç¨ä¸ä¸ªæµç¨å¾å°±å¯ä»¥è§£é
æ´æ°å
ç±äºkeyçé»è®¤å¼ä¸ºnullï¼æ以æ´æ°åä¸æ´æ°å满足keyç¸åä¸å ç´ ç±»åä¸åï¼é£ä¹æ们è¦å é¤æ´æ°åçä¸ä¸ªdivèç¹ï¼æ°å¢pèç¹
ä¸ãå¤èç¹diff
对äºå¤èç¹diff,æ们è¦éånewChildrenåoldFiberè¿è¡æ¯è¾ãç±äºReactå¢éåç°domèç¹ä¸è¬ææ´æ°ï¼å¢å ï¼å é¤è¿ä¸ç§æä½ï¼èæ´æ°æ´ä¸ºé¢ç¹ï¼æ以ä»ä»¬è®¾ç½®æ´æ°çä¼å 级é«äºå¢å å é¤ãåºäºä»¥ä¸åå ï¼å¨å¤èç¹diffç®æ³çå®ç°ä¸æ两å±éåï¼ç¬¬ä¸å±éåå¤çæ´æ°çèç¹ï¼ç¬¬äºå±éåå¤çæ´æ°ä»¥å¤çèç¹ã
第ä¸å±éå
éånewChildrenä¸oldFiber,å¤æèç¹æ¯å¦å¯å¤ç¨ï¼å¦æå¯ä»¥å¤ç¨ï¼å继ç»éåã
å¦æä¸è½å¤ç¨ï¼å为两ç§æ åµï¼
第äºå±éå
第äºå±éåä»ç¬¬ä¸å±éåçç»æä½å¼å§
第ä¸å±éåç»æåæ4ç§ç»æ
é¦å æ们è¦å¤ænewChildrenä¸éåå°çèç¹ï¼å¨oldFiberä¸æ¯å¦åå¨ï¼åºäºæ¤ï¼Reactå°oldFiberä¸çèç¹ä»¥key-oldfiberé®å¼å¯¹çå½¢å¼åå¨Mapä¸ï¼åªéè¦newChildrençkeyï¼å°±å¯ä»¥å¤æoldFiberä¸æ没æç¸åºçèç¹ã
å¦æoldFiberä¸æ²¡æç¸åºçèç¹ï¼åå°newChildrençæçfiberæä¸placementæ è®°
å¦ææç¸åºçèç¹ï¼å°å®çç´¢å¼è®°ä¸ºoldIndexï¼ä¸ä¸ä¸æ¬¡å¯å¤ç¨èç¹å¨oldFiberçç´¢å¼ä½ç½®lastPlacedIndexæ¯è¾ï¼å¦ææ¯æ¬¡å¯å¤ç¨çèç¹å¨ä¸ä¸æ¬¡å¯å¤ç¨å³è¾¹å°±è¯´æä½ç½®æ²¡æååï¼å³
è¥oldIndex=lastPlacedIndex,说æç¸å¯¹ä½ç½®æ²¡æååï¼é£ä¹ä»¤lastPlacedIndex=oldIndex
è¥oldIndexlastPlacedIndex,代表æ¬èç¹éè¦åå³ç§»å¨ã
ä¾å¦:
åèææ¡£ï¼
Reactææ¯æç§(iamkasong.com)
Reactdiffç®æ³reactä½ä¸ºä¸æ¬¾æ主æµçå端æ¡æ¶ä¹ä¸ï¼å¨è®¾è®¡çæ¶åé¤äºç®åæä½ä¹å¤ï¼æ注éçå°æ¹å°±æ¯èçæ§è½äºãdiffç®æ³å°±æ¯ä¸ºèçæ§è½è设计çï¼diffç®æ³åèæDOMçå®ç¾ç»åæ¯reactææé åçå°æ¹ãå ¶ä¸ï¼diffæ¯differentçç®åï¼è¿æ ·ä¸æ¥ï¼diffç®æ³æ¯ä»ä¹ä¹å°±é¡¾åæä¹äºââæ¾ä¸åã
å¨DOMéè¦æ´æ°çæ¶åï¼éè¿diffç®æ³å¯ä»¥è®¡ç®åºèæDOMä¸çæ£ååçé¨åï¼ä»èåªé对ååçé¨åè¿è¡æ´æ°æ¸²æï¼é¿å âçµä¸åèå¨å ¨èº«âï¼é ææ§è½æµªè´¹ã
è½ç¶å®ç¾å°å®ç°äºæ¾ä¸åçåè½ï¼ä½æ¯å»çå¼ç循ç¯éå½å¯¹èç¹è¿è¡ä¾æ¬¡ç对æ¯ï¼ä½¿å ¶ç®æ³çæ¶é´å¤æ度为O(n^3)ï¼å ¶ä¸næ¯domæ çèç¹æ°ãå¦ædomæ°è¶³å¤å¤§çè¯ï¼è¿ä¸ªç®æ³å°å¯¹cpuå½¢æç»æã
为äºä¼ådiffç®æ³ï¼reactä¸å¯¹æ®éçdiffç®æ³å®è¡äºä¸å¤§çç¥ï¼æåå°æ¶é´å¤æ度é为O(n)
treediffæ¯diffç®æ³çåºç¡çç¥ï¼å®çéç¹å¨äºåå±æ¯è¾ã
åºäºå¯¹diffç®æ³çä¼åï¼reactçtreediff对DOMèç¹çè·¨å±çº§ç§»å¨çæä½å¿½ç¥ä¸è®¡ï¼react对VirtualDOMæ è¿è¡å±çº§æ§å¶ï¼ä¹å°±æ¯è¯´åªå¯¹ç¸åå±çº§çDOMèç¹è¿è¡æ¯è¾ï¼å³åä¸ä¸ªç¶èç¹ä¸çææåèç¹ï¼ã对æ¯æ¶ï¼ä¸æ¦åç°èç¹ä¸åå¨ï¼åç´æ¥å é¤æ该èç¹ä»¥åä¹ä¸çææåèç¹ãè¿æ ·ç§©åºå¯¹DOMæ è¿è¡ä¾æ¬¡éåï¼å°±å¯ä»¥å®ææ´ä¸ªæ ç对æ¯ãæ¶é´å¤æ度为O(n)
ä¸ä¸ªçé®ï¼æ¢ç¶treediff忽ç¥äºè·¨å±çº§ç§»å¨çæä½ï¼å¦æè¿ç§æ åµåºç°äºï¼diffç®æ³ä¼æä¹å¤çå¢ï¼
çï¼ä¸ç®¡ï¼å¤äºå°±æ°å¢ï¼å°äºå°±å é¤ï¼åªæå建èç¹åå é¤èç¹çæä½ï¼ãæ以å®æ¹æ确建议ä¸è¦è¿è¡DOMèç¹çè·¨å±çº§æä½ã
componentdiffæ¯ç»ä»¶é´ç对æ¯
å¨éå°ç»ä»¶ä¹é´çæ¯è¾æ¶ï¼æä¸ç§çç¥
ä¼åç¹ï¼
elementdiffæ¯é对åä¸å±çº§çelementèç¹ç
å¨åæ¹åä¸å±çº§çèç¹å¯¹æ¯æ¶ï¼æä¸ç§æ åµ
åèç¹æ´æ°æ¶ï¼ä¼åºç°ä»¥ä¸å ç§æ åµï¼
reactä¸çkeyå¼ï¼å®ä¸æ¯ç»å¼åè 使ç¨çãå¨ä¸è¬æ åµä¸keyå¼æ¯å½æ们å¨ååå ç´ éåçæ¶åéè¦ä½¿ç¨çãå 为æ们å¦æè¦è¿è¡æ°æ®çæ´æ°ï¼å°±éè¦è¿è¡èædomç对æ¯ï¼èkeyå¼å°±æ¯æ¯ä¸ªå ç´ èç¹å¯¹åºçå¯ä¸å¼ãè¿ä¸ªæ¶åå°±éè¦å¯¹æ¯åå ç´ çkeyå¼æ¯å¦æå¹é 项ï¼å¦ææçæ åµä¸åä¼è¿è¡æ°æ®çæ´æ°ï¼å¦ækeyå¼æ²¡æå¹é 项ï¼é£ä¹è¿ä¸ªèç¹å°±éè¦è¿è¡å é¤åéæ°å建ã
å æ¤æ们å¨éåçæ¶ååä¸ä¸è¦ç¨indexä¸æ æè æ¶é´æ³ãéæºæ°çè¿è¡keyå¼çèµå¼ãè¿æ ·ä¼é æå ç´ ç移é¤éæ°å建浪费æ§è½ã
reactå¤èç¹diffç®æå®ç°
reactæ¯ä¸ä¸ªæ°æ®é©±å¨çæ¡æ¶ï¼éè¿å°æ°æ®ä¸UIå ³èèµ·æ¥è¾¾å°æ°æ®æ´æ°æ¶åæ¶æ´æ°UIæ´æ°çç®çã对äºreactwebappæ¥è¯´ï¼æ°æ®çåå¨æç»ä¼è½¬å为domçååãå½ç¶react并ä¸ä¼å¯¹domè¿è¡ç´æ¥æ¯è¾ï¼èæ¯å¯¹æ¯åååçfiberã对fiberçdiffæç»ä¼åæ å°domä¸ã
å å设å¨fiberååæ¶ä¸ä½¿ç¨diffç®æ³ï¼å³ä¸æ¦fiberæ¹ååå é¤åååçææfiber并æå ¥åååçfiberãè¿ç§æ¹æ³è½ç¶ç®ä¾¿ï¼ä½åå¨æ§è½é®é¢ï¼å 为domçå é¤åå建é½éè¦èè´¹æ¶é´ãä¾å¦ï¼fiberä»a,b,cå为a,c,bãåªéè¦å°bæå ¥å°cä¹åå³å¯ï¼æ éå建任ä½fiberãå æ¤ï¼éè¦ä¸ç§æ¹æ³æ¥æ è®°å ç´ çåæ´ï¼è¿å°±æ¯diffç®æ³ã
å¦æåååé½åå¨å¤ä¸ªå ç´ ï¼åå±äºå¤èç¹çdiffãå¤èç¹çfiberdiff对äºæ¯ä¸ä¸ªfiberå®é åªåå¨ä¸¤ç§æ åµ:
为ä»ä¹ç§»å¨ææ°å¢domé½å±äºåä¸ç§æ åµï¼å 为reactå®é ä¸æç»ä¼è°ç¨Node.insertBefore()æ¥è¿è¡placementæä½ï¼å ¶å®ä¹å¦ä¸:
å æ¤react并ä¸å ³å¿è¯¥fiberæ¯ç§»å¨(å·²ç»åå¨)è¿æ¯æ°å¢(ä¸åå¨éè¦å建)ãä¾å¦fiberä»a,b,c,då为a,c,b,dï¼é£ä¹reactä¼å°bè¿ä¸ªfiberæ 记为Placementãå ¶ä½fiberä¸åãå¨æç»è¿è¡domååæ¶è°ç¨parent.insertBefore(d,b)ãå æ¤diffçç®ç并ä¸æ¯è¦ä¸¥æ ¼çæ¾åºfiberä»åªä¸ªä½ç½®ç§»å¨å°åªä¸ªä½ç½®ï¼åªéè¦å¾åºåªäºéè¦å é¤ï¼åªäºéè¦Placementå³å¯ã
å设åå¨now以åbefore两个fiberéåã为äºç®ååºæ¯ï¼è®¤ä¸ºnowä¸çfiberå¨beforeä¸é½åå¨ãè¿æ¶åé®é¢å¯ä»¥è½¬æ¢ä¸ºå¦ä½ç§»å¨beforeä¸çå ç´ å°å ¶è½¬æ¢ä¸ºnowãreactå¤çåæ³ä¸ºå³ç§»beforeä¸çé¨åfiberå°å ¶è½¬æ¢ä¸ºnowãä¾å¦ï¼before以åafterä¸keyç顺åºä¸º:
é£ä¹æ è®°b为Placementå³å¯ã对äºè¿ä¸ªä»»å¡ï¼æ们å°ä¸ä¸ä¸ªä½ç½®ä¸åçå ç´ å¨nowä¸çä½ç½®è®°ä¸ºlastKeepIndexï¼å½éånowæ°ç»ä¸çæ¯ä¸ªfiberæ¶ï¼å¦æ该fiberå¨beforeæ°ç»ä¸åå¨ï¼ä¸ãå说æå½åæéåå°å¾fiberå¨:
è¿å°±æå³è¿è¿ä¸ªfiberæ¯éè¦ç§»å¨çãå¦æä¸æ»¡è¶³è¿ä¸ªæ¡ä»¶ï¼åéè¦è¯¥fiberç¸å¯¹lastKeepIndexææ è®°çfiberä½ç½®æ²¡æåå¨ï¼æ éæ¹åã
å½ç¶ï¼å®é ä¸ä¸å¯è½nowä¸çfiberå¨beforeä¸é½è½æ¾å°ãä½è¿ç§åæ ·ç´æ¥æ 记为Placementå³å¯ãåæ¶å¨beforeä¸å´ä¸å¨nowä¸çéè¦å ç´ æ 记为Deletionã为äºæ¹ä¾¿è¿éæ们å®ä¹4ç§ç±»åçDiff:
æ´ä¸ªdiffçé»è¾ä¸º:
å¨å¾å°diffçç»æåï¼reactéè¿ä¸¤ä¸ªdomæä½å½æ°æ¥å°diffåºç¨å°çå®çdom:
第ä¸ä¸ªå½æ°å¯¹åºäºåååéè¦è¿è¡Placementæå å¼èç¹çæ åµï¼ä¾å¦fiberä»a,b,c,dåå为a,c,b,dãæ¤æ¶b被æ 记为Placementãreactä¼æ¾å°åååå®ç第ä¸ä¸ªä¸éè¦åå¨çå å¼èç¹å³ä¸ºdï¼å¹¶è°ç¨parent.insertBefore(d,b)ãå®æåçå®çdomå°±ä»a,b,c,dåæa,c,b,dã
第äºä¸ªå½æ°å¯¹åºäºåååéè¦è¿è¡Placementä¸åå¨å å¼èç¹çæ åµï¼ä¾å¦fiberä»a,b,cåå为a,c,bæ¤æ¶b被æ 记为Placementï¼ä½å ¶ä¸åå¨å å¼èç¹ãreactä¼è°ç¨parent.appendChild(b)ãå®æåçå®çdomå°±ä»a,b,cåæa,c,bã
å½ç¶ï¼çå®çæ åµæ¯è¿è¦æ´å¤æãå æ¤æå ¥domå¿ å®è¦å æ¾å°fiberæ ä¸çæ£çdomèç¹ãèfiberæ å®é ä¸æ¯ç¨æ·èªå®ä¹ç»ä»¶fiber以åçå®domfiberç»åå¨ä¸èµ·çï¼å¦ä½æ¾å°çå®çå å¼domèç¹å¯¹åºçfiberä¹æ¯ä¸ä¸ªæ¯è¾å¤æçä»»å¡ã
reactéè¿diffç®æ³æ¥è¿è¡æ§è½ä¼åï¼åå°domçå建åå é¤ãé£ä¹reactéç¨çä¼åæ¯å¦ä¸ºæä¼åå¢ï¼çæ¡æ¯ï¼å¦ãä¾å¦åå¨è¿æ ·ä¸ä¸ªç¹æ®çä¾åï¼
ç±äºreactdiffç®æ³çå±éï¼è¿ééè¦å°1ä»ç§»å¨å°ä¹åï¼ä½å®é ä¸æ们ä¸ç¼å°±è½çåºæç®åçæ¹æ³æ¯å°ç§»å¨å°1ä¹åãè¿ä¹å°±æ¯æè¿å¾å¤æ¡æ¶å¼å§ä½¿ç¨æé¿ä¸åååºåæ¥ä¼ådiffç®æ³çåå ãé£ä¹é®é¢æ¥äºï¼ä½ ç¥é为ä»ä¹è¿éreactéè¦ç§»å¨ä¸ªå ç´ ï¼æè 说为ä»ä¹æé¿ä¸åååºåå¯ä»¥è§£å³æ´ä¸ªé®é¢å?
è¡ä»£ç å®ç°Reactæ ¸å¿Diffç®æ³è¯¥å¦ä½è®¾è®¡Diffç®æ³å¢ï¼èèå°åªæ以ä¸ä¸ç§æ åµï¼ä¸ç§å¸¸è§ç设计æè·¯æ¯ï¼
æè¿ä¸ªæ¹æ¡ï¼å ¶å®æ个éå«çåæââä¸åæä½çä¼å 级æ¯ç¸åçãä½å¨æ¥å¸¸å¼åä¸ï¼èç¹ç§»å¨åçè¾å°ï¼æ以Diffç®æ³ä¼ä¼å å¤æå ¶ä»æ åµã
åºäºè¿ä¸ªç念ï¼ä¸»æµæ¡æ¶ï¼ReactãVueï¼çDiffç®æ³é½ä¼ç»åå¤è½®éåï¼å å¤ç常è§æ åµï¼åå¤çä¸å¸¸è§æ åµã
æ以ï¼è¿å°±è¦æ±å¤çä¸å¸¸è§æ åµçç®æ³éè¦è½ç»åç§è¾¹çcaseå åºã
æ¢å¥è¯è¯´ï¼å®å ¨å¯ä»¥ä» 使ç¨å¤çä¸å¸¸è§æ åµçç®æ³å®æDiffæä½ã主æµæ¡æ¶ä¹æ以没è¿ä¹åæ¯ä¸ºäºæ§è½èèã
æ¬æä¼ç æå¤ç常è§æ åµçç®æ³ï¼ä¿çå¤çä¸å¸¸è§æ åµçç®æ³ã
è¿æ ·ï¼åªéè¦è¡ä»£ç å°±è½å®ç°Diffçæ ¸å¿é»è¾ã
é¦å ï¼æ们å®ä¹èæDOMèç¹çæ°æ®ç»æï¼
keyæ¯nodeçå¯ä¸æ è¯ï¼ç¨äºå°èç¹å¨åååãåååå ³èä¸ã
flag代表nodeç»è¿Diffåï¼éè¦å¯¹ç¸åºççå®DOMæ§è¡çæä½ï¼å ¶ä¸ï¼
index代表该nodeå¨å级nodeä¸çç´¢å¼ä½ç½®
注ï¼æ¬Demoä» å®ç°ä¸ºnodeæ è®°flagï¼æ²¡æå®ç°æ ¹æ®flagæ§è¡DOMæä½ã
æ们å¸æå®ç°çdiffæ¹æ³ï¼æ¥æ¶æ´æ°åä¸æ´æ°åçNodeListï¼ä¸ºä»ä»¬æ è®°flagï¼
æ¯å¦å¯¹äºï¼
{ key:"d",flag:"Placement"}代表d对åºDOMéè¦æå ¥é¡µé¢ã
{ key:"a",flag:"Deletion"}代表a对åºDOMéè¦è¢«å é¤ã
æ§è¡åçç»æå°±æ¯ï¼é¡µé¢ä¸çaå为dã
åæ¯å¦ï¼
ç±äºbä¹åå·²ç»åå¨ï¼{ key:"b",flag:"Placement"}代表b对åºDOMéè¦åå移å¨ï¼å¯¹åºparentNode.appendChildæ¹æ³ï¼ãabcç»è¿è¯¥æä½åå为acbã
ç±äºaä¹åå·²ç»åå¨ï¼{ key:"a",flag:"Placement"}代表a对åºDOMéè¦åå移å¨ãacbç»è¿è¯¥æä½åå为cbaã
æ§è¡åçç»æå°±æ¯ï¼é¡µé¢ä¸çabcå为cbaã
æ ¸å¿é»è¾å æ¬ä¸æ¥ï¼
æ们å°beforeä¸æ¯ä¸ªnodeä¿åå¨ä»¥node.key为keyï¼node为valueçMapä¸ã
è¿æ ·ï¼ä»¥O(1)å¤æ度就è½éè¿keyæ¾å°beforeä¸å¯¹åºnodeï¼
å½éåafteræ¶ï¼å¦æä¸ä¸ªnodeåæ¶åå¨äºbeforeä¸afterï¼keyç¸åï¼ï¼æ们称è¿ä¸ªnodeå¯å¤ç¨ã
æ¯å¦ï¼å¯¹äºå¦ä¸ä¾åï¼bæ¯å¯å¤ç¨çï¼
对äºå¯å¤ç¨çnodeï¼æ¬æ¬¡æ´æ°ä¸å®å±äºä»¥ä¸ä¸¤ç§æ åµä¹ä¸ï¼
å¦ä½å¤æå¯å¤ç¨çnodeæ¯å¦ç§»å¨å¢ï¼
æ们ç¨lastPlacedIndexåéä¿åéåå°çæåä¸ä¸ªå¯å¤ç¨nodeå¨beforeä¸çindexï¼
å½éåafteræ¶ï¼æ¯è½®éåå°çnodeï¼ä¸å®æ¯å½åéåå°çæænodeä¸æé å³çé£ä¸ªã
å¦æè¿ä¸ªnodeæ¯å¯å¤ç¨çnodeï¼é£ä¹nodeBeforeä¸lastPlacedIndexåå¨ä¸¤ç§å ³ç³»ï¼
注ï¼nodeBefore代表该å¯å¤ç¨çnodeå¨beforeä¸ç对åºnode
代表æ´æ°å该nodeå¨lastPlacedIndex对åºnode左边ã
èæ´æ°å该nodeä¸å¨lastPlacedIndex对åºnode左边ï¼å 为ä»æ¯å½åéåå°çæænodeä¸æé å³çé£ä¸ªï¼ã
è¿å°±ä»£è¡¨è¯¥nodeåå³ç§»å¨äºï¼éè¦æ è®°Placementã
该nodeå¨åå°ï¼ä¸éè¦ç§»å¨ã
ç»è¿éåï¼å¦æbeforeMapä¸è¿å©ä¸nodeï¼ä»£è¡¨è¿äºnode没æ³å¤ç¨ï¼éè¦è¢«æ è®°å é¤ã
æ¯å¦å¦ä¸æ åµï¼éåå®afteråï¼beforeMapä¸è¿å©ä¸{ key:'a'}ï¼
è¿æå³çaéè¦è¢«æ è®°å é¤ã
æ以ï¼æåè¿éè¦å å ¥æ è®°å é¤çé»è¾ï¼
Minimap2 用户手册
Minimap2是一个高效快速的序列比对工具,专门用于处理长读段数据,如PacBio或Oxford Nanopore基因组读取。它能够映射长读段或组装到参考基因组,并提供详细比对选项。Minimap2以PAF或SAM格式输出结果。主要功能包括:成对映射(默认输出格式):PAF格式,每行至少包含个字段,用于显示映射位置。
限制:在长低复杂性区域,可能产生次优比对,因种子位置可能不理想。
编译要求:需要SSE2或NEON指令集,可选不支持以减慢程序速度。
Minimap2适用于多种应用场景,如:映射长噪声读段,处理人类基因组等大型数据库。
查找读段间的重叠。
剪接感知比对,包括PacBio Iso-Seq、Nanopore cDNA或RNA数据。
比对Illumina短读段。
组装比对。
两个物种的全基因组比对,差异度低于%。
性能优势:处理噪声读取序列时,Minimap2的速度远超主流映射器。
对于kb以上序列,性能显著优于BLASR、BWA-MEM、NGMLR和GMAP。
在长读取映射上更准确,比对具有生物学意义,适合后续分析。
对于Illumina短读取,Minimap2速度更快,准确性与BWA-MEM和Bowtie2相当。
安装与使用:预编译二进制文件可从发布页面获取。
从源代码编译需安装C编译器、GNU make和zlib开发文件。
支持SIMD Everywhere (SIMDe)库实现移植,适用于不同SIMD指令集。
可无缝处理gzip压缩的FASTA和FASTQ格式输入。
构建参考数据库的最小化索引,加速映射过程。
使用选项调整参数以优化性能和准确性。
使用案例与参数调整:选择预设选项以获得最佳性能和准确性。
映射长噪声基因组读取时,调整参数以匹配数据类型。
映射长mRNA/cDNA读取时,使用特定选项加快比对速度,提高准确性。
通过基因组注释优化比对过程。
调整剪接参数以适应不同数据类型。
高级功能与限制:处理>个CIGAR操作的SAM格式,可能需要选项-L将长CIGAR移动到CG标签。
可选的cs标签编码不匹配和INDEL处的碱基信息,便于后续分析。
Minimap2附带的paftools.js脚本用于处理PAF格式比对并提供评估工具。
详细算法概览和开发者指南提供API文档,支持C和Python接口。
限制在长低复杂性区域可能产生次优比对。
总的来说,Minimap2是一个功能丰富、性能高效的序列比对工具,适用于多种大规模数据比对任务,提供灵活的参数调整以适应不同数据类型和需求。MAFFT:序列比对软件linux版下载安装
Mafft是一款用于序列比对的软件,支持MacOSX、Linux和Windows操作系统。在进行序列比对前,建议关注序列的方向,尽管Mafft软件是否具备自动反向互补功能尚不明确。
对于Linux系统的用户,Mafft提供了rpm和dpkg两种安装方式。同时,您也可以选择从源代码安装。具体步骤如下:首先,下载文件“mafft-7.-with-extensions-src.tgz”,此版本支持RNA结构比对以及protein、DNA、RNA序列比对功能。如需仅进行protein、DNA、RNA序列比对,可以选择下载“mafft-7.-without-extensions-src.tgz”。
请注意,安装Mafft时可能需要root权限,对于普通用户而言,参照相关教程进行安装。在安装过程中,需修改Makefile文件以指定安装路径。Mafft将被安装到当前目录的core下,并在您设定的路径下的bin目录中建立链接。
为了了解Mafft的使用方法及其参数,可以通过执行“./mafft --help”命令获取帮助信息。这将提供详细的使用指南,帮助您高效地进行序列比对操作。